alphafold2のTwitterイラスト検索結果。 13 件
A #MachineLearning method, AlphaFold2, "predicts protein structures with near-experimental accuracy."
I read again with pleasure about this breakthrough!
#Science
#AI
@emblebi
⏯️Protein-structure prediction revolutionized
1-https://t.co/4r1fmqRXRc
2-https://t.co/LKXDQpJOGf
/AlphaFold2の非専門家向け活用法 第4回前半「AlphaFoldが現状カバーできていない構造情報」|上海老師|note https://t.co/GNnuOGbV6n
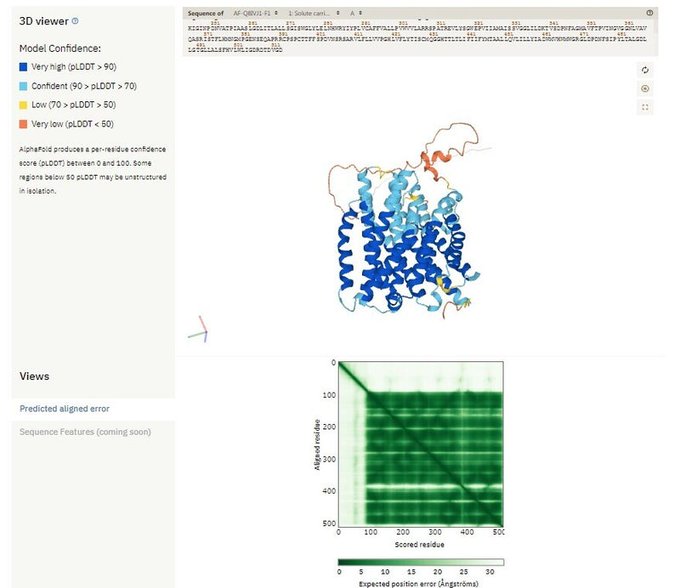
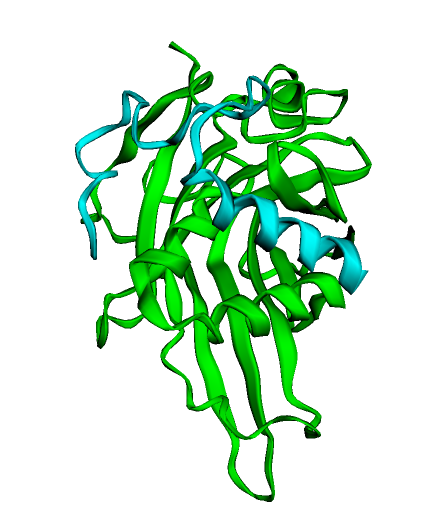
遅ればせながら、ようやく時間ができたのでAlphaFold2で遊んでる。ATeamで使ってる枯草菌のFoF1-εの配列入れて見たところ、リガンドのATP無くてもC末端ヘリックスバンドルが折りたたまれた結果が出てきた。これは予想外。
AlphaFold2すげーな。Drosophila melanogasterのscallopedタンパクA鎖の予測させたらほぼ正解だった。なお、素人が遊んでるだけなので全然科学的ではない。
左、AlphaFold2の予測 右、既知の立体構造
https://t.co/2YHKFVz9JA
#AlphaFold2
世界最小10残基のタンパク質ことシニョリン(chignolin)の熱安定変異体CLN025で試したらかなり結晶構造とずれていた。1枚目:結晶構造(5awl), 2枚目: 予測構造, 3,4枚目: 重ね合わせ RMSD=1.45
https://t.co/wSdzBFZahq
AlphaFold2で構造が既知の蛍光タンパク質をいくつか試してみたが、発色団周りは全然合致せず。Gamillusの発色団はトランス体のp-hydroxybenzylidene-imidazolidinoneだけど、AlphaFold2の予測構造は発色団ができていない。これでは光異性化やESPTなどの予測もできない・・・。
タンパク質ってこんな形してるんだ👀
タンパク質の構造を解析するAI「AlphaFold2」が無料公開!
東京大学の教授が「6年間解けなかった分子の結晶構造があっさり解けた」とツイート✨✨
下記で誰でもAlphaFold2で解析できる
(上から▶️を順番に押すだけでOK😀)
https://t.co/pRgg2qDYuN
#alphafold2
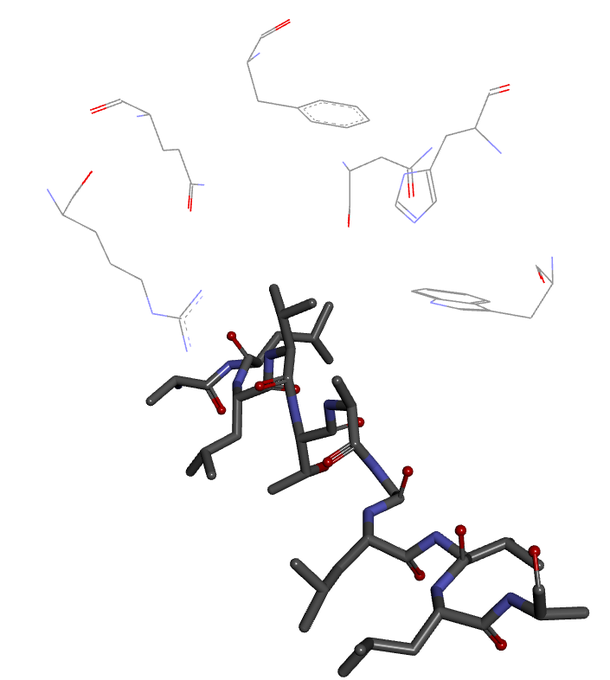
簡易版AF2使って、CyclosporinとCyclophilinAの複合体構造(1CWH)で遊んでみた。左が実測、右が予測。
近いところに寄って行ってはいるのだが……。流石に環状ペプチドは厳しいのかな
#AlphaFold #AlphaFold2
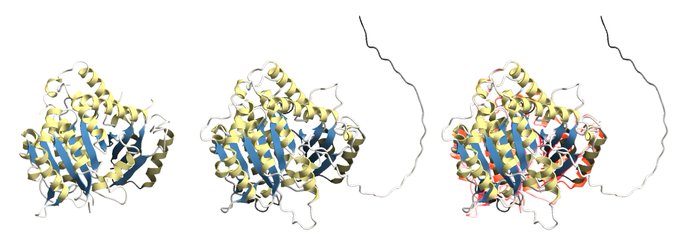
Alphafold2ではペプチド以外扱えない為、apo体(水色)の構造が出てほしいところだが、リファレンスは錯体結合型の構造を多く使用する為、holo型の構造が出てしまうのも、問題点なのかな。(構造は緑膿菌のヘム輸送タンパクHasA、ピンク色:自分と縁があるコバルトテトラフェニルポルフィセン結合型)
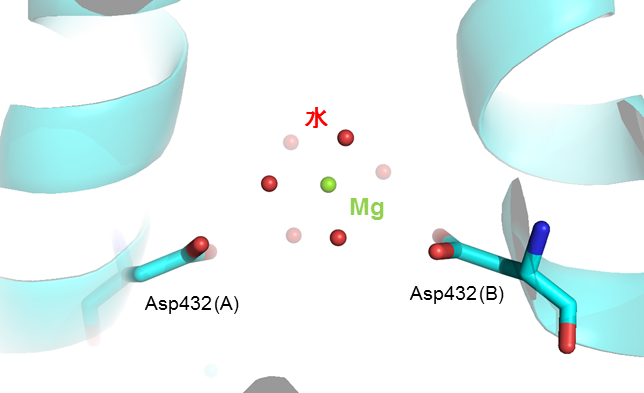
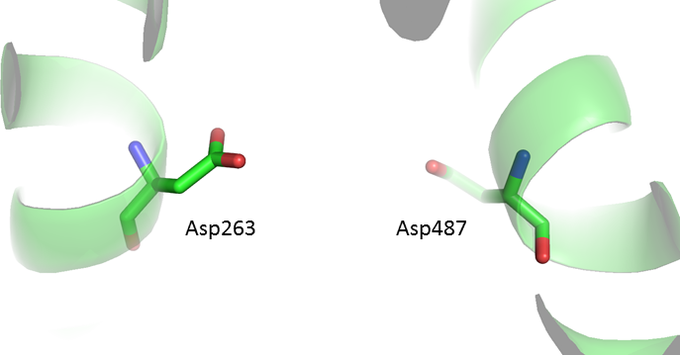
AlphaFold2で予測した構造は、活性部位付近の側鎖の配向もほぼ正解に限りなく近いレベルかなと思いました。画像だとちょっとよく伝わらないかもしれませんが。